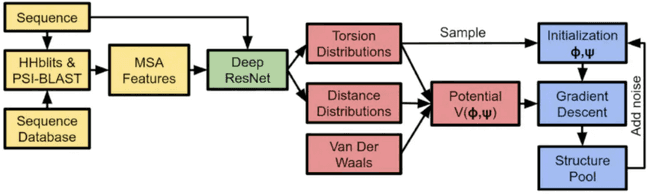
若说2021年人工智能学术界最令人振奋的成果,那么Alphafold可谓当之无愧。Alphafold2在CASP14 蛋白质预测挑战上取得了远远超出同类模型的准确率,并首次将蛋白质结构预测的精度提高到了原子级别——已经接近了实验测量的水准。
幻方AI团队在Alphafold2推出不久就在萤火二号平台成功将Alphafold2训练运行了起来,详情如我们上一期文章《萤火跑模型 | Alphafold 蛋白质结构预测》。然而,由于Alphafold2本身模型的复杂性和开源代码的缺陷,并且幻方AI的训练平台也比较特殊,使得一开始复现资源利用效率非常底下,训练成本不可接受。
为了提高Alphafold的训练效率,帮助科研人员与开发者们降低研究门槛,幻方AI团队对Alphafold2模型进行了大量优化,大幅提高了模型训练的效率。我们的工作可以被概括为以下三点:
- 重构代码,解决开源版本中无法多机多卡训练的问题,利用数据并行大幅提高Alphafold2训练效率,降低复现门槛;
- 优化数据处理逻辑,大幅增加GPU的利用率,从40%左右提升到了90%以上;
- 利用hfai高性能工具改造模型,将Alphafold2深度融合进幻方AI的训练平台中,大幅提高训练性能。
本次文章先和大家聊聊,如何优化蛋白质数据的处理,以获得高效的训练性能。
模型仓库:https://github.com/HFAiLab/alphafold-optimized
输入优化
Alphafold的输入特征可以分为这样三类:
- 蛋白质序列本身
- Multi-Sequence Alignment(MSA)多序列对齐特征
- Template蛋白质模板特征
它们在不同的蛋白质序列长度之间差异非常大,处理所需时间可以从几秒到上千秒不等,这会极大的影响模型训练的效率。例如在一次20分钟的训练过程中,单个长样本的处理就可能阻塞整体训练10几分钟。而在多机多卡大批次场景下,每个批次内有部分数据的处理时间极长几乎成了必然事件。
如上图所示,在训练Alphafold的初始阶段,输入的特征是通过序列数据库搜索,生物学工具(JackHmmer/HHSearch/…)计算而得出,需要依赖大量的CPU计算能力支持。在整个Alphafold模型训练的过程中,CPU算力往往成为了阻碍Alphafold训练性能提升的重要瓶颈。
针对上述问题,幻方AI采用特征预处理与特征裁切两种方案对输入进行优化。简单来说,特征预处理利用专门的CPU集群,将大量数据库搜索任务,生物学工具计算任务预先做掉,得到特征集和候选集;在模型训练阶段,模型直接拉取特征集和候选集,筛选特征,适当裁切长蛋白质序列特征。通过该方法我们实现了alphafold训练性能4-5倍的提升,在接下来的章节中我们会为大家详细描述。
1. 特征预处理
在一般的模型训练任务中,耗时的大头都在于GPU的前向推理和反向传播的计算,数据处理可以通过异步Dataloader载入的方式来掩盖时间开销。然而在Alphafold的训练中,模型对处理好的数据进行一次迭代需要大约11秒(基于A100 GPU),而对于这一个数据的预处理则需要花费大约150秒(一整颗EPYC 64核处理器),其是GPU运算时间的13倍,根本无法被GPU计算时所隐藏。 在这种情况下,GPU利用率的理论上限也仅有7.3%,效率是非常低下的。 同时,萤火二号集群上每个机器配备的是8张GPU和一张CPU,按照原有的多卡并行训练方式,CPU资源会更加紧缺,因此在训练时对特征处理进行优化已经毫无意义,我们需要寻找其他替代方案。
我们选择将特征处理单独分离出来,使用CPU集群算力预先完成一部分任务,将得到的候选样本数据进行保存。为了提高处效率,最大化CPU的利用率,我们使用Python多进程对特征预处理进行了并行加速。然而,特征处理涉及到多个不同的生物学工具,且每种工具对CPU的利用率各不相同,因此直接并行化很容易导致进程过多,从而产生大量的CPU调度抢占开销。根据统计,在对几个不同类型的特征进行处理时的CPU资源占用情况如下表所示:
| 蛋白质数据集 | 处理工具 | 耗时(秒) | CPU占用核数 | 总算力需求(分钟/核) |
|---|---|---|---|---|
| Uniref90 | JackHmmer | 567 | 4核 | 37.80 |
| Magnify | JackHmmer | 851 | 4核 | 56.73 |
| Bfd-small | JackHmmer | 308 | 4核 | 20.53 |
| Pdb70 | HHSearch | 62 | 64核 | 66.13 |
从上表可以观察到:前三个数据集使用JackHmmer,其基本只能使用4核的CPU资源;而Pdb70使用HHSearch,其能够使用CPU上的所有资源。因此我们需要针对不同的处理工具建立不同的进程池进行调度分配。我们采用的优化策略是:同时发起多个JackHmmer任务,并每隔一段时间发起一个HHSearch任务。
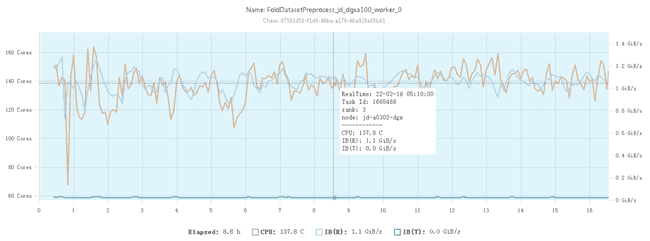
在128核的CPU节点上,经过优化后的特征预处理的CPU占用率如下图所示:
可见此时已经可以基本以比较稳定的100%利用率使用CPU资源。
值得注意的是,特征预处理可以预先将蛋白质本身特征提取出来(序列信息、空间结构信息等),而对于MSA特征和Template特征,Alphafold的论文中提到,训练中是一个搜索过程,随机选取最合适的4个特征进行输入,因此预处理能提供适合适的候选集,而最终模型训练时选哪4个特征作为输入,预处理部分无法替代。
上述特征预处理出来的中间结果可以通过幻方AI自研的FFRecord格式进行存储,以充分利用萤火集群的高速文件系统3FS,详情可以参考之前的文章《幻方力量 | 高速文件系统:3FS》。
2. 特征裁切
前面提到,不同长度的蛋白质序列处理可能会导致训练被阻塞,除了将运行时处理转为预处理,我们还可以对较长的特征进行裁切。但需要注意的是,对特征进行特征裁切必然会影响模型训练的效果,因此需要在模型性能和训练效率二者间调节平衡。
特征裁切仅针对MSA特征和Template特征的处理,因为其主要目标是找到更多相似特征(序列、结构)以增强模型的表达能力。特征裁切可以有效减少对复杂蛋白质的特征处理开销,提高整个训练效率。蛋白质序列本身特征是不适用特征裁切的。
2.1 MSA特征裁切
MSA特征是一组与输入的人类蛋白质序列相似的其他生物蛋白质序列的集合,用于帮助模型Encoder部分形成蛋白质的特征向量。若输入的蛋白质序列长度为N个氨基酸,在预处理阶段中找到的同源蛋白质数量为M,则需要进行预处理的MSA特征的大小就为NxM。可见在蛋白质序列不能被裁切的前提下,只能通过对同源蛋白质数量进行裁切,即丢弃部分之前查找到的同源序列来减少这部分的耗时。
蛋白质序列长度N,MSA特征序列数M,最终处理时长T,他们满足如下线性关系:
C是一个固定常数,和机器的处理能力有关。我们实验中发现,使训练不被阻塞的最大时长为150秒,因此我们可以根据上面的公式推得每个输入蛋白质序列的最大MSA特征数量。实际在实验中,只有不到10%的序列需要裁切MSA特征,因此这能够在尽可能不损害性能的情况下极大的提升训练的效率。
2.2 Template特征裁切
Template特征旨在抽取与要预测的蛋白质在空间结构上相似的蛋白质序列,用于帮助模型的Structure Module部分形成特征表达,输出蛋白质的空间结构。同样地,单个蛋白质可能有上百个模板,虽然在Alphafold的训练中最终只有4个模板需要被送入模型参与训练,但未必所有模板都与蛋白质真正匹配。因此一旦遇到大量模板都不符合要求的情况,在检查模板序列的过程中就可能要花费十几分钟的时间。
对于Template特征的处理可以进行并行加速,但受限于PyTorch Dataloader守护进程的问题和CPU资源量的限制,在实际训练中这种方法不太适用。因此我们同样考虑对Template特征进行裁切,以减少这步骤消耗的时间。
- 特征预处理获得Template特征候选集,按照匹配度进行排序;
- 训练时尽量使用与原蛋白质更匹配的模板;
- 对于模板匹配过程设有一个固定的时间限制,超时做裁切。
后面实验会展示上述方法在实际训练中的效果。
实验
首先,我们先测试Alphafold在单张A100显卡上进行训练时每一个Step的时间开销,作为基准:
| Stage | Mean duration (s) | Percentage (%) |
|---|---|---|
| Training Step | 12.65 | 100 |
| Get Training Batch | 1.87 | 14.78 |
| Forward | 4.24 | 33.52 |
| Zero Grad | 0.17 | 1.31 |
| Exponential Moving Average | 0.14 | 1.0 |
| Optimizer Step | 0.45 | 3.42 |
| Backward | 5.93 | 46.88 |
从表格中可以看到,训练一个Step就要花费12.65秒,其中最主要的时间开销在于Backward,Forward和Get Training Batch三步。由于使用的数据很少,并且没有使用多卡并行,此时特征处理带来的性能瓶颈和DDP的开销还没有能体现出来。
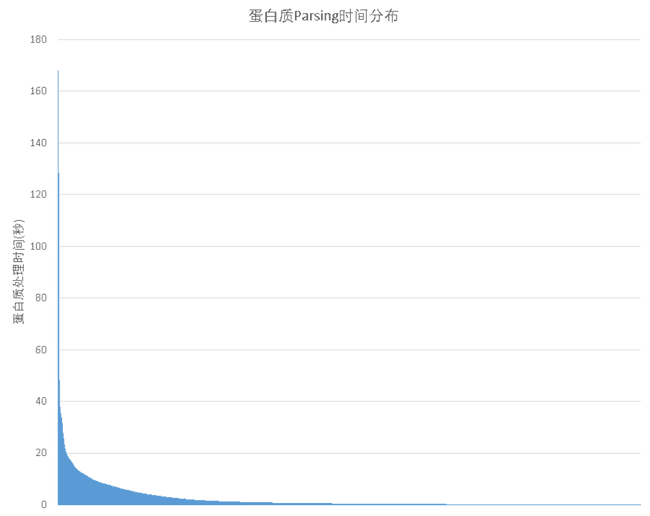
而当训练数据较多时,不同训练数据间的处理时间差异就会显得尤为突出。如下图所示,大多数输入的数据只需要数秒即可完成处理,然而少部分蛋白质在极端情况下会需要上百秒的预处理时间。
因此一旦遇到一条这样的输入数据,就会导致整个训练阻塞。而假设所有训练数据中只有5%会导致训练阻塞,那么在使用128的batch_size训练Alphafold(原论文参数)时,每个batch被阻塞的概率就为:
这几乎在每个step的训练中都会遇到阻塞的情况。此时整个训练的GPU利用率会很极端的下滑到20%以 下,其中大部分时间都花费在了等待batch中的某一个数据处理完成上。
例如在没有进行优化时使用16个节点,128张A100进行训练,那么前4个step的花费时间就为:413s, 177s, 100s, 101s, 每个step中的GPU利用率不到10%。可见此时若是不对数据处理进行优化,那么很难正常进行训练。
1. 特征预处理测试
我们在总共11万条的蛋白质序列训练数据中,随机抽取了3.5万条蛋白质序列进行解析,并统计了这些序列分别使用直接训练时处理和特征预处理方式的耗时:
| 蛋白质 | 原处理时间(s) | 使用特征预处理后的时间(s) |
|---|---|---|
| 3j3q | 145.9 | 46.6 |
| 3j3y | 129.8 | 41.0 |
| 6u42 | 78.9 | 27.5 |
如上表所示,对于处理时长大于10秒的序列,原来需要150秒的特征解析过程在优化后仅仅需要50秒左右,这大大抑制了超长序列对整个训练的阻塞影响。即使是小序列,也能得到将近3倍的速度提升。
接着,我们再看看使用特征预处理结果对训练时的GPU利用率和总训练时间能带来多大的收益。这里,我们先不进行特征裁切,先比较特征预处理方法后的性能提升。结果如下表所示:
| 不进行特征预处理 | 进行特征预处理 | |
|---|---|---|
| 每轮开始时的数据处理时长(s) | 451 | 393 |
| 平均每个step时长(s) | 50.3 | 23.57 |
| 训练被阻塞的次数 | 10 | 6 |
| GPU利用率 | 19.58% | 42.37% |
可以看到,进行特征预处理能够获得非常明显的性能提升,GPU利用率提升了一倍。但同时,过多的训练阻塞导致整体的GPU利用率依然不够理想。
2. MSA特征裁切测试
接着,我们测试使用MSA特征裁切对训练时的GPU利用率和总训练时间的收益。这里MSA特征裁切时间常数为 2e-6,不进行Template特征裁切,都使用特征预处理。结果如下表所示:
| 不进行MSA裁切 | 裁切MSA特征 | |
|---|---|---|
| 每轮开始时的数据处理时长(s) | 393 | 200 |
| 平均每个step时长(s) | 23.57 | 10.87 |
| 训练被阻塞的次数 | 6 | 1 |
| GPU利用率 | 42.37% | 90.2% |
可以看到,使用MSA特征裁切后GPU利用率能再提升一倍以上,训练阻塞也只发生一次。该方法能有效提升训练效率。
3. Template特征裁切测试
之后,我们再测试下Template特征裁切对训练效率的提升效果。时间限制为150秒,都使用特征预处理,不进行MSA特征处理。结果如下表所示:
| 不进行Template裁切 | 裁切Template特征 | |
|---|---|---|
| 每轮开始时的数据处理时长(s) | 393 | 201 |
| 平均每个step时长(s) | 23.57 | 11.65 |
| 训练被阻塞的次数 | 6 | 1 |
| GPU利用率 | 42.37% | 90.2% |
使用Template特征裁切后GPU利用率同样能够提升一倍以上。
小结
作为2021年AI领域最具影响力的工作之一,Alphafold的复现无疑是有意义的,能帮助更多研究者和开发者们去探索这个方向的前景。通过优化数据输入,我们可以实现90%以上的GPU使用率,高效利用起了集群计算资源。同时,幻方AI也很多高效的训练工具,比如hfreduce,3FS,算子等等,它们是否能使得Alphafold的训练进一步加速?下一期技术分享将为大家呈现。